Nature | MD安德森癌症中心开发新型单细胞测序方法,证实乳腺肿瘤在进展过程中保持亚克隆多样性
Nature | MD安德森癌症中心开发新型单细胞测序方法,
证实乳腺肿瘤在进展过程中保持亚克隆多样性

非整倍体是人类乳腺癌的一个显著特征,在携带TP53突变的三阴性乳腺癌(TNBC)患者中尤其普遍。目前,科学家在模型系统中已经阐明了非整倍体的基本分子机制,但在人类原发性肿瘤的生长过程中,人们对染色体重排的了解仍然有限。肿瘤进展的普遍范例是,突变和染色体畸变随着时间的推移逐渐积累,进而导致癌症恶性阶段。除此之外,还存在间断拷贝数进化(PCNE)模式,其中许多染色体重排是在肿瘤进化早期,在基因组不稳定的短期爆发中一起获得的。该模式已在乳腺癌、结肠癌和前列腺癌中被报道。
在基因组不稳定性的初始爆发后,是否还存在持续的拷贝数变化?此前研究发现,PCNE在TNBC7患者中很常见,但无法确定在肿瘤细胞进行克隆扩增后,拷贝数图谱是否继续进化。由于可测序的细胞数量有限,且第一代单细胞DNA测序(scDNA-seq)技术存在广泛的技术噪音,因此解析该类模型非常困难。
为研究这一过程,美国得克萨斯大学安德森癌症中心科研团队开发了一种单细胞、单分子DNA测序方法。利用该方法,研究团队对来自8种人类三阴性乳腺癌和4种细胞系的16178个单细胞进行了测序及拷贝数分析,解决了有关TNBC患者染色体进化自然史的基本问题。该项研究近日以“Breast tumours maintain a reservoir of subclonal diversity during expansion”为题发表在Nature上。

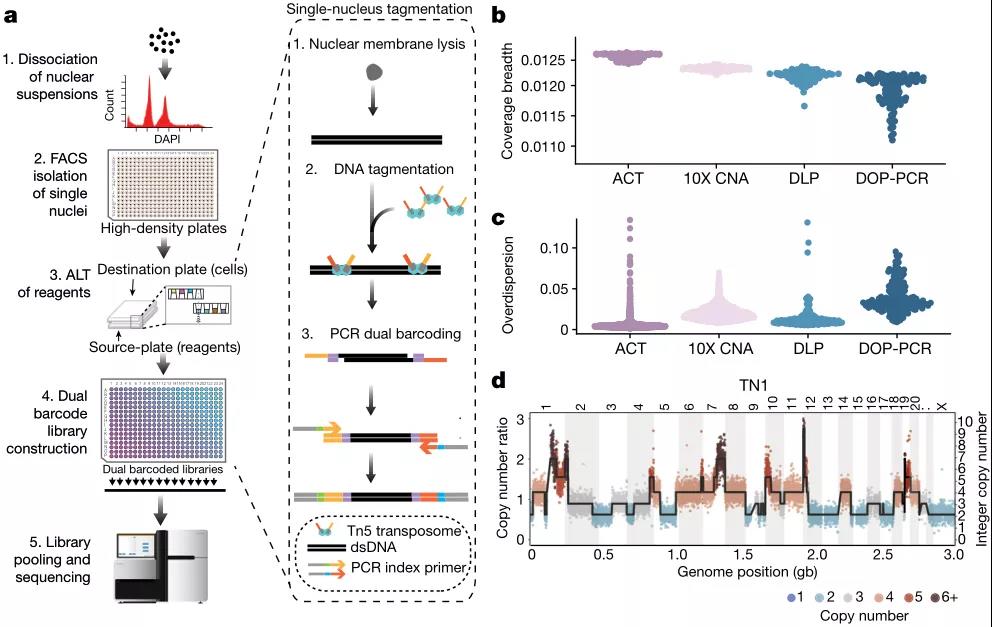
研究者通过结合单核的荧光激活细胞分选(FACS)、标记和声学液体转移(ALT)技术,开发了一种声学细胞标记法(ACT),以单分子分辨率进行高通量scDNA-seq。,研究者利用新鲜或冷冻组织制备核悬液,并将分离的细胞核经过自动化的三步扩增化学过程,生成平均大小为312bp的条形码单细胞DNA库,这些DNA库被汇集在一起用于下一代测序。
与依赖全基因组扩增步骤的第一代scDNA-seq方法相比,ACT所需实验步骤少,时间短,通量高且检测单分子DNA信息的能力强。研究团队评估了该方法的覆盖广度和技术噪声,结果显示ACT比其他三种scDNA-seq方法(10X CNV、DLP、DOP-PCR)都有显著的改善(P<0.05);且ACT单细胞的覆盖均匀性与批量DNA测序数据相似,比DOP-PCR数据更均匀。因此ACT实现了对现有scDNA-seq方法的技术改进(图1)。
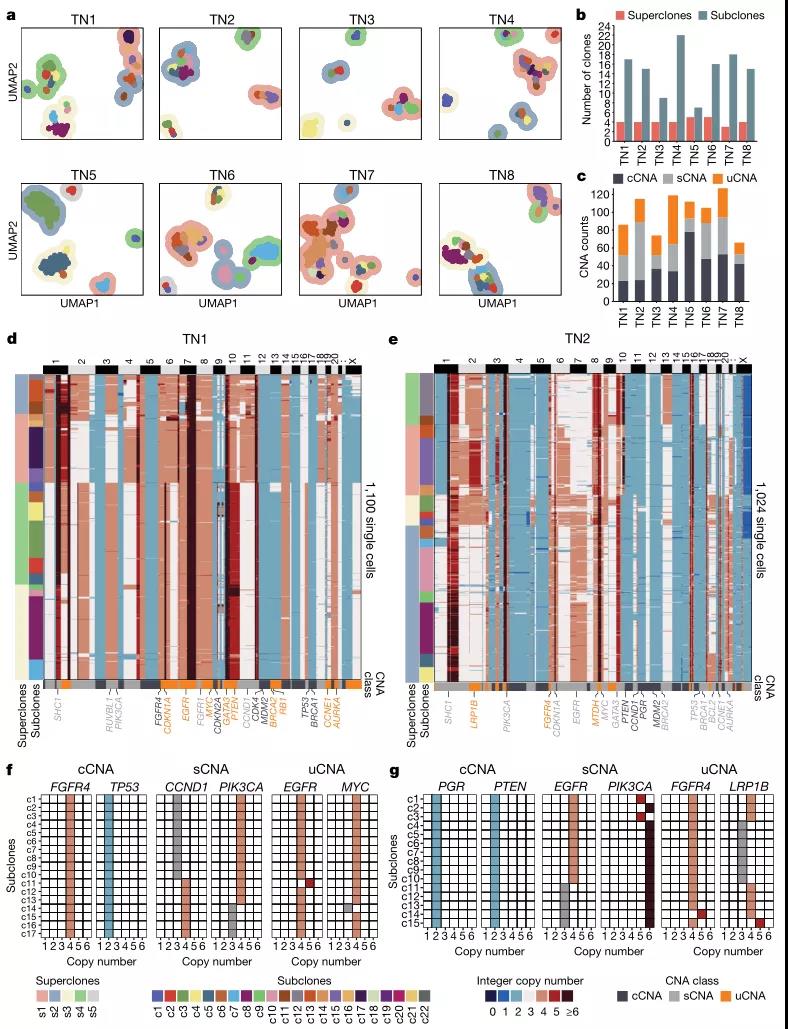
研究人员利用ACT对来自8个TNBC肿瘤的9,765个细胞进行了测序,包括TN1 DCIS样本、3个未经治疗的浸润性导管癌(TN2、TN6和TN7)以及4个未经治疗的同步DCIS-IDC样本(TN3-TN5和TN8)。经过聚类分析,共识别出7~22个亚克隆,这些亚克隆被组织成横跨8个肿瘤的3~5个超克隆。结果显示,TN3和TN5的亚克隆数量最低,其余肿瘤的亚克隆数量和基因组多样性指数较高。拷贝数变异(CNA)分类因肿瘤而异,TN5的克隆性CNA(cCNA)事件最多,TN4的唯一CNA(uCNA)计数最高。在TN1样本中,单细胞数据显示17个亚克隆被分成4个超克隆,超克隆中许多事件与乳腺癌基因交叉。上述研究表明,乳腺肿瘤和细胞系由大量亚克隆(7-22个)组成,这些亚克隆被组织成3-5个主要的超克隆(图2)。
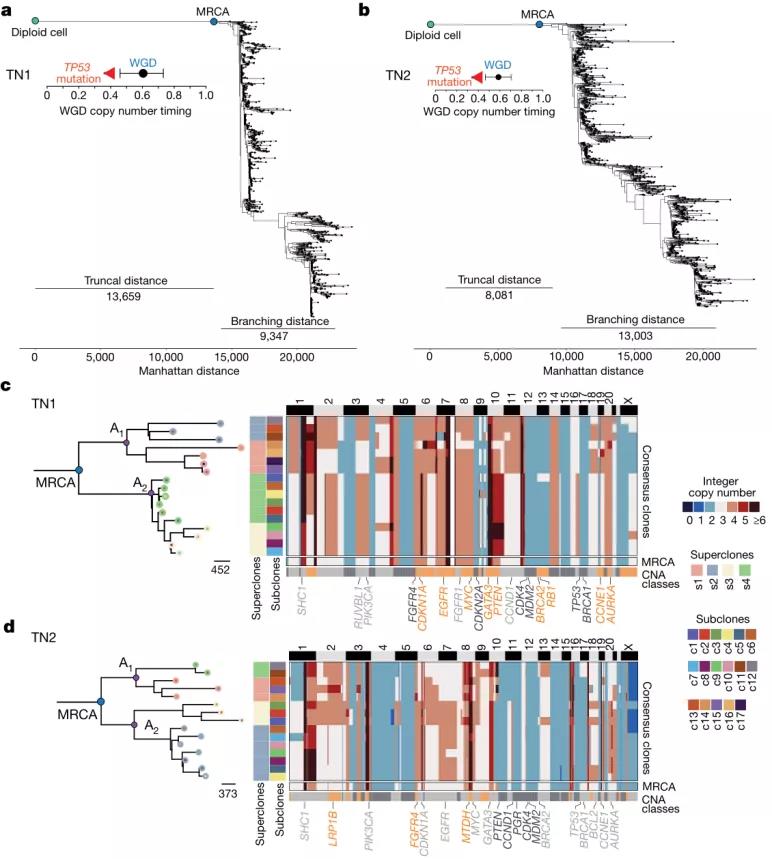
接下来,研究团队重建了原发肿瘤扩展前CNAs的进化,对8种肿瘤和匹配的正常组织进行外显子组测序,结果显示有平均102个体细胞突变,包括所有肿瘤中的TP53驱动突变。为了研究最近共同祖先序列(MRCA)后的肿瘤进化,研究团队使用ACT数据推断系统发育树,结果显示大量的CNAs是克隆性的,在MRCA之后,所产生的树显示出较大距离的分支谱系,且8种肿瘤在MRCA后拷贝数仍在进化。以上数据表明三阴性乳腺癌在MRCA后获得了大量的sCNA和uCNAs,在原发肿瘤肿块的扩展过程中,继续使克隆基因型多样化(图3)。
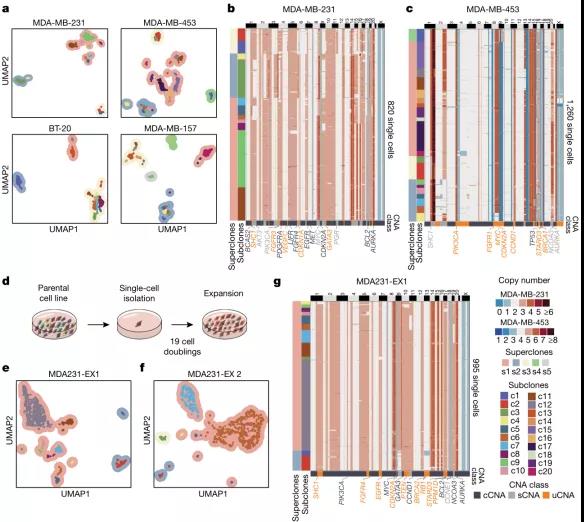
此外,研究团队还分析了在人类TNBC肿瘤中观察到的广泛拷贝数多样性是否也存在于TNBC细胞系中。该研究选择了4个具有TP53突变和非整倍体核型(MDA-MB-231,BT-20,MDA-MB-157和MDA-MB-453)的TNBC细胞系,应用ACT对6413个细胞进行测序,然后使用聚类分析描绘其克隆性亚结构。结果显示,与原发肿瘤相似,4种细胞系有11-20个亚克隆,分成3-5个超克隆;cCNA、sCNA和uCNA事件的Shannon多样性指数和频率与TNBC肿瘤相似,片段大小分布也相似。总体而言,测序数据表明这些细胞系代表了人类TNBC肿瘤的拷贝数亚结构。同时研究发现,单个癌细胞在扩增后,即使在相对较短的时间内,也不能保持稳定的克隆基因型。并进一步证实亚克隆CNAs事件除了对基因剂量有局部影响外,还对整个基因组中许多基因在通路和癌症标志物中的表达有更广泛的影响(图4)。
综上所述,该研究报道了一种单细胞、单分子DNA测序方法——ACT,利用该方法证实了三阴性乳腺癌在原发肿瘤生长过程中继续进化染色体畸变,并保持亚克隆多样性。与第一代scDNA-seq方法相比,ACT在技术性能上有了改进,且性价比高,在生物学和生物医学等领域研究非整倍体方面将有广泛的应用。
来源:https://mp.weixin.qq.com/s/wFZ1X2tE1Poore5YAkRCsw

